Анализ выдачи программы PROCHECK для записи 1NZJ PDB
Карта рамачандрана:

Процент остатков (отличных от Gly и Pro), попавших в предпочитаемые области на карте - 93.6%:No. of residues %-tage ------ ------ Most favoured regions [A,B,L] 220 93.6%Структура с таким значением (>90%) считается хорошей.Significant regions (Остатки с RSR>10%)
В моем белке 16 остатков (6%) с пространственным R-фактором больше нормы (10%). У некоторых из таких остатков, например, Asn223 (RSR=0.325) и Lys240 (RSR=0.410), недостает отдельных атомов (об этом сказано в поле REMARK) или они находятся рядом с неполными остатками. У N-концевого (из отраженных в структуре) Thr4 также большой RSR (0.312).
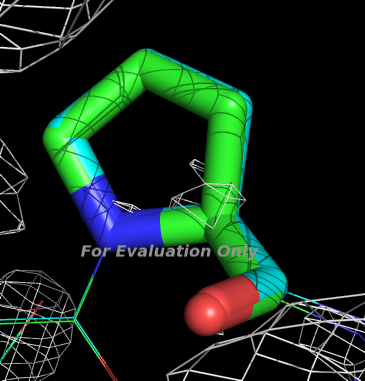
Для описания я выбрала Pro239 (RSR=0.409). Изображение остатка и экспериментальной электронной плотности:
Уровень
электронной
плотностиИзображение 1.5 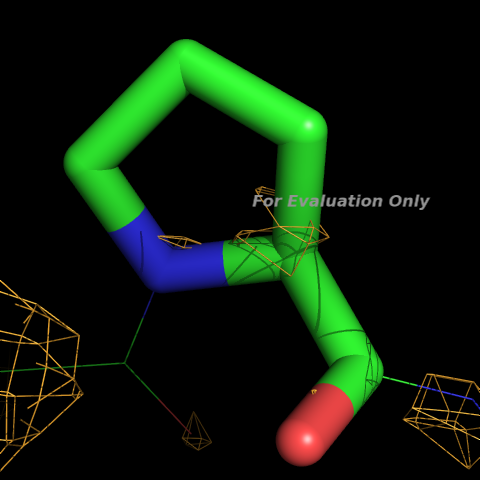
1.0 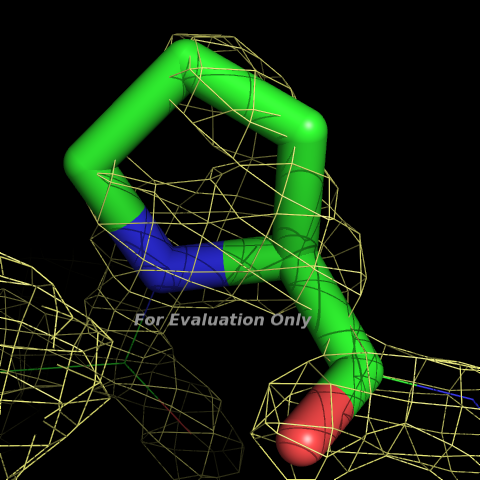
0.5 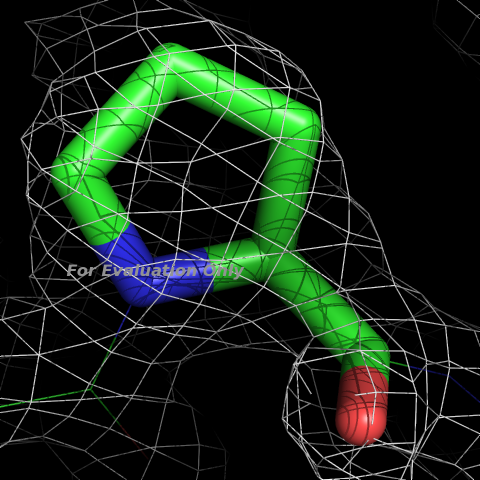
Т. е. для этого остатка зафиксирована пониженная электронная плотность.PDB_REDO
У оптимизированной структуры:
R-values- улучшились значения sigmaR-free, R-free Z-score
- ухудшились значения R и R-free
WHAT_CHECK validation- улучшились значения 1st и 2nd generation packing quality, Chi-1/Chi-2 rotamer normality, Backbone conformation,
- ухудшились значения Ramachandran plot appearance, Bond length и angle RMS Z-score (хотя осалось <1), Total number of bumps,
Анализ оптимизированной структуры.
Как уже было замечено выше, в большинстве остатков с плохим RSR отсутствуют некоторые атомы. В оптимизированной структуре они восстановлены, например, в Asn223 добавлены недостающие атомы CG, OD1 и ND2 (на картинке оптимизированная структура - бирюзовая):
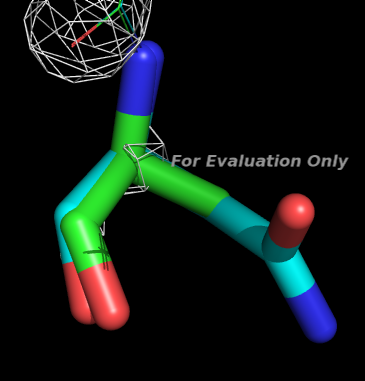
Правда, остатки с плохими значениями RSR не стали лучше вписываться в электронную плотность, т. к. проблема была в самой плотности. Разобранный выше Pro239: