Отобранные бактерии
Название Мнемоника Rhizobium etli RHIEC Ralstonia pickettii RALPJ Erwinia carotovora ERWCT Salmonella typhimurium SALTY Yersinia pseudotuberculosis YERPS Vibrio fischeri VIBFM Proteus mirabilis PROMH Скобочная формула дерева
(RHIEC, (RALPJ, (VIBFM, (PROMH, (YERPS, (ERWCT, SALTY))))));
Изображение дерева
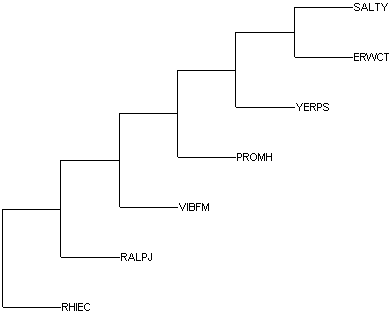
Ветви дерева
Дерево содержит четыре нетривиальные ветви:
1) {RHIEC, RALPJ} против {VIBFM, PROMH, YERPS, ERWCT, SALTY}
2) {RHIEC, RALPJ, VIBFM,} против {PROMH, YERPS, ERWCT, SALTY}
3) {RHIEC, RALPJ, VIBFM, PROMH} против {YERPS, ERWCT, SALTY}
4) {RHIEC, RALPJ, VIBFM, PROMH, YERPS} против {ERWCT, SALTY}
Занятие 2.
- Таксоны выбранных бактерий (определены с помощью таксономического сервиса NCBI:
Название Таксон Rhizobium etli Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Rhizobium Ralstonia pickettii Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia Erwinia carotovora Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Pectobacterium Salmonella typhimurium Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Salmonella; Salmonella enterica; Salmonella enterica subsp. enterica Yersinia pseudotuberculosis Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia Vibrio fischeri Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Aliivibrio Proteus mirabilis Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Proteus
Ветвь {VIBFM, PROMH, YERPS, ERWCT, SALTY} выделяет Гамма-протеобактерии.
Ветвь {PROMH, YERPS, ERWCT, SALTY} выделяет энтеробактерии.
-
Выбранная функция: Энолаза (ENO)
Файл с последовательностями белков ENO_RHIEC, ENO_RALPJ, ENO_VIBFM, ENO_PROMH, ENO_YERPS, ENO_ERWCT, ENO_SALTY в fasta-формате.
-
Выравнивание отобранных белков программой muscle:
файл msf
файл fasta
*Диагностические позиции выравнивания
Импортированное в GeneDoc выравнивание (выделены консервативные для группы гаммапротеобактерий позиции. Для энтеробактерий позиции те же, другую группу составить нельзя):

Диагностические позиции выравнивания - позиции, в которых все последовательности из данного таксона содержат некоторый остаток, не встречающийся в этой позиции в последовательностях, не относящихся к таксону. Диагностические позиции: K3, V5, K6, A23, E24, H26, F31, M/L34, A35, V71, G73, P74, A76, K85, D86, F106, A124, K126, H133, I134, L135, N138, T140, S145, E158, N163, Q172, K180, A194, V196, K198, K200, A220, E228, A232, L237, K239, T242, K268, F270, T271, E274,H277, Q285, Q306, D312, F345, K359, D363, A380, A390, A417, P423, N425, K428, E429, K431, Q433.
-
Реконструкция дерева программой fprotpars.
Программа выдала 3 дерева. Скобочные формулы:(((RALPJ,RHIEC),(((YERPS,ERWCT),SALTY),PROMH)),VIBFM)[0.3333]; ((((RALPJ,RHIEC),((YERPS,ERWCT),SALTY)),PROMH),VIBFM)[0.3333]; ((((RALPJ,RHIEC),(YERPS,(ERWCT,SALTY))),PROMH),VIBFM)[0.3333];
Изображения:
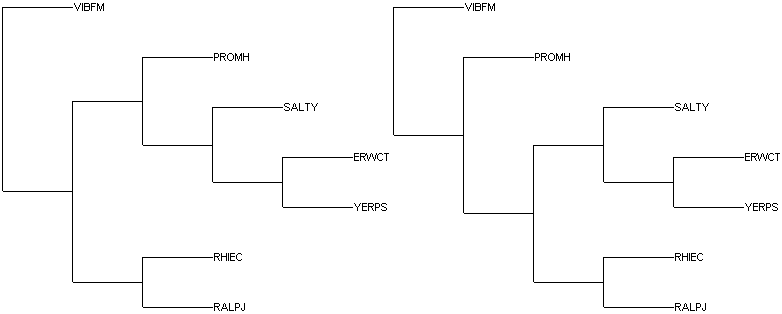
Сравним первое из полученных деревьев с правильным. Его нетривиальные ветви:
1){RALPJ, RHIEC} против {YERPS, ERWCT, SALTY, PROMH, VIBFM}
2){RHIEC, RALPJ, VIBFM,} против {PROMH, YERPS, ERWCT, SALTY}
3){RHIEC, RALPJ, VIBFM, PROMH} против {YERPS, ERWCT, SALTY}
4){RHIEC, RALPJ, VIBFM, PROMH, SALTY} против {ERWCT, YERPS}
Разбиения 1, 2 и 3 у деревьев совпадают, но 4-ые различные: у правильного дерева есть ветви {RHIEC, RALPJ, VIBFM, PROMH, YERPS} против {ERWCT, SALTY}, но нет {RHIEC, RALPJ, VIBFM, PROMH, SALTY} против {ERWCT, YERPS}.
-
Оценка эволюционных расстояний между последовательностями программой fprotdist.
Матрица расстояний:VIBFM PROMH SALTY ERWCT YERPS RHIEC RALPJ VIBFM 0.000000 0.152839 0.142313 0.143594 0.121746 0.525515 0.447445 PROMH 0.152839 0.000000 0.095445 0.106934 0.106114 0.526616 0.457518 SALTY 0.142313 0.095445 0.000000 0.053870 0.063430 0.484270 0.435582 ERWCT 0.143594 0.106934 0.053870 0.000000 0.041740 0.507924 0.431272 YERPS 0.121746 0.106114 0.063430 0.041740 0.000000 0.500986 0.426479 RHIEC 0.525515 0.526616 0.484270 0.507924 0.500986 0.000000 0.435659 RALPJ 0.447445 0.457518 0.435582 0.431272 0.426479 0.435659 0.000000Аддитивность: если есть четыре последовательности A,B,C,D, то из трех сумм
1) d(A,B) + d(C,D) 2) d(A,C) + d(B,D) 3) d(A,D) + d(B,C)
две равны между собой и больше третьей.
Возьмем VIBFM, ERWCT, YERPS и RALPJ.
1)0,143594+0,426479=0,568384
2)0,121746+0,431272=0,553018
3)0,447445+0,041740=0,489185
1 и 2 различаются на 0,015366, больше 3-ей.
'Ультраметрическое пространство': d (A,B) <= max(d (A,C), d (B,C))
Для PROMH, SALTY и YERPS: 0.095445 <= max(0.106114, 0.063430) - верно (меньше на 0,010669)
Для VIBFM, RHIEC и RALPJ: 0.525515 <= max(0.447445, 0.435659) - не верно (больше на 0,07807)
Реконструкция дерева программой fneighbor.
Результаты действия двух алгоритмов (слева Neighbor-Joining, справа UPGMA):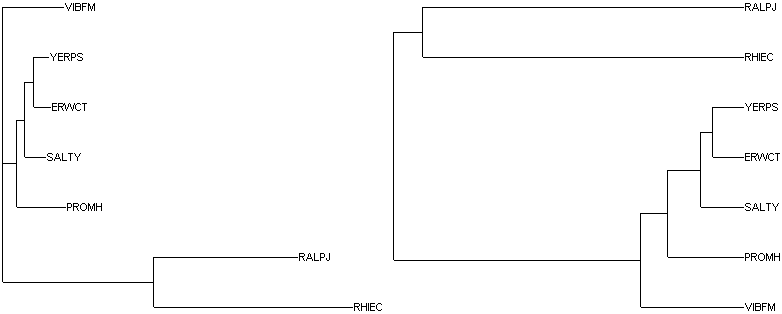
Скобочные формулы:
((RHIEC:0.25253,RALPJ:0.18313):0.19101,(PROMH:0.06152,(SALTY:0.02692,(ERWCT:0.02172,YERPS:0.02002):0.01086):0.01014):0.01769,VIBFM:0.07764);
((VIBFM:0.07006,(PROMH:0.05142,(SALTY:0.02932,(ERWCT:0.02087,YERPS:0.02087):0.00845):0.02209):0.01865):0.16712,(RHIEC:0.21783,RALPJ:0.21783):0.01935);
Приведенные деревья имеют одинаковые нетривиальные ветви, но отличаются ими от настоящего: нет ветви {RHIEC, RALPJ, VIBFM, PROMH} против {YERPS, ERWCT, SALTY} и, соответственно, {RHIEC, RALPJ, VIBFM, PROMH, YERPS} против { ERWCT, SALTY}. У деревьев различны длины ветвей. Алгоритм UPGMA выдает укорененное дерево, хотя на изображении, созданном TreeDyn этого не видно. Дерево, построенное через Neighbor-Joining - явно не ультраметрическое.