Главная страница
Реконструкция филогенетических деревьев
1. Скобочная формула, рисунок дерева
Исходная скобочная формула -
((((А:40,(В:16,С:16):24):10,D:50):40,Е:90):10,F:100);
С помощью Paint по заданной формуле строим следующее дерево:
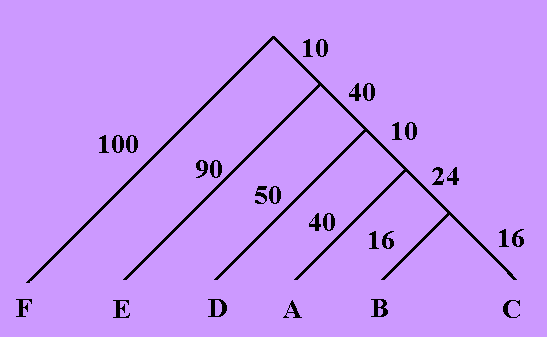
2. Описание ветвей дерева как разбиения множества листьев
Исходя из полученного дерева и считая его бескорневым, составляем таблицу ветвей (опущены ветви, соответствующие одному листу).
| A
|
B
|
C
|
D
|
E
|
F
|
| .
|
*
|
*
|
.
|
.
|
.
|
| *
|
*
|
*
|
.
|
.
|
.
|
| *
|
*
|
*
|
*
|
.
|
.
|
3. Получение искусственных мутантных последовательностей
Длина гена fabI - 789 нуклеотидов.
Формула для пересчета расстояний в число мутаций в гене:
(x/100)*789, где х - число замен на 100 нуклеотидов (эволюционное расстояние, полученное из заданной скобочной формулы).
Скрипт для получения мутантных последовательностей
msbar fabI.fasta F.fasta -point 4 -count 789 -auto
msbar fabI.fasta ABCDE.fasta -point 4 -count 79 -auto
msbar ABCDE.fasta E.fasta -point 4 -count 710 -auto
msbar ABCDE.fasta ABCD.fasta -point 4 -count 316 -auto
msbar ABCD.fasta D.fasta -point 4 -count 394 -auto
msbar ABCD.fasta ABC.fasta -point 4 -count 79 -auto
msbar ABC.fasta BC.fasta -point 4 -count 189 -auto
msbar ABC.fasta A.fasta -point 4 -count 315 -auto
msbar BC.fasta B.fasta -point 4 -count 126 -auto
msbar BC.fasta C.fasta -point 4 -count 126 -auto
cat F.fasta>>align.fasta
cat E.fasta>>align.fasta
cat D.fasta>>align.fasta
cat A.fasta>>align.fasta
cat B.fasta>>align.fasta
cat C.fasta>>align.fasta
После выполнения скрипта выравнивание мутантных последовательностей находится в файле align.fasta
4) Реконструкция дерева алгоритмами максимального правдоподобия, Neighbor-joining и UPGMA.
1. Реконструкция дерева методом максимального правдоподобия
Выполняем команду
fdnaml align.fasta -ttratio 1 -auto
Полученное дерево:
+------------------------E
|
| +---C
| +----4
| +----3 +---B
| | |
1----2 +---------A
| |
| +--------D
|
+---------------------------------F
2. Реконструкция дерева методом Neighbor-joining
Рассчитываем попарные расстояния между последовательностями программой fdnadist:
fdnadist align.fasta -ttratio 1 -auto
Подаем результаты на вход программе fneighbor:
fneighbor align.fdnadist -auto
Полученное дерево:
+------------------------E
!
! +--------D
3----4
! ! +----------A
! +----2
! ! +----B
! +----1
! +---C
!
+----------------------------------F
2. Реконструкция дерева методом UPGMA
Еще раз подаем результаты расчета расстояний между последовательностями на вход программе fneighbor, но с параметром -treetype u
fneighbor align.fdnadist -treetype u -auto
Полученное дерево:
+----------------------------------------------------F
--5
! +------------------------------------------E
+---------4
! +---------------------D
+--------------------3
! +------------------A
+--2
! +-------B
+----------1
+-------C
Сравниваем полученные деревья:
| A
|
B
|
C
|
D
|
E
|
F
|
Алгоритм максимального правдоподобия
|
Neighbor-joining
|
UPGMA
|
Правильное дерево
|
| .
|
*
|
*
|
.
|
.
|
.
|
+
|
+
|
+
|
+
|
| *
|
*
|
*
|
.
|
.
|
.
|
+
|
+
|
+
|
+
|
| *
|
*
|
*
|
*
|
.
|
.
|
+
|
+
|
+
|
+
|
Как можно увидеть, ветви деревьев полностью идентичны. Различия в расположении листьев относительно друг друга обусловлены алгоритмом реконструкции дерева.