Моделирование и реконструкция эволюции гена
Моделирование и реконструкция эволюции гена
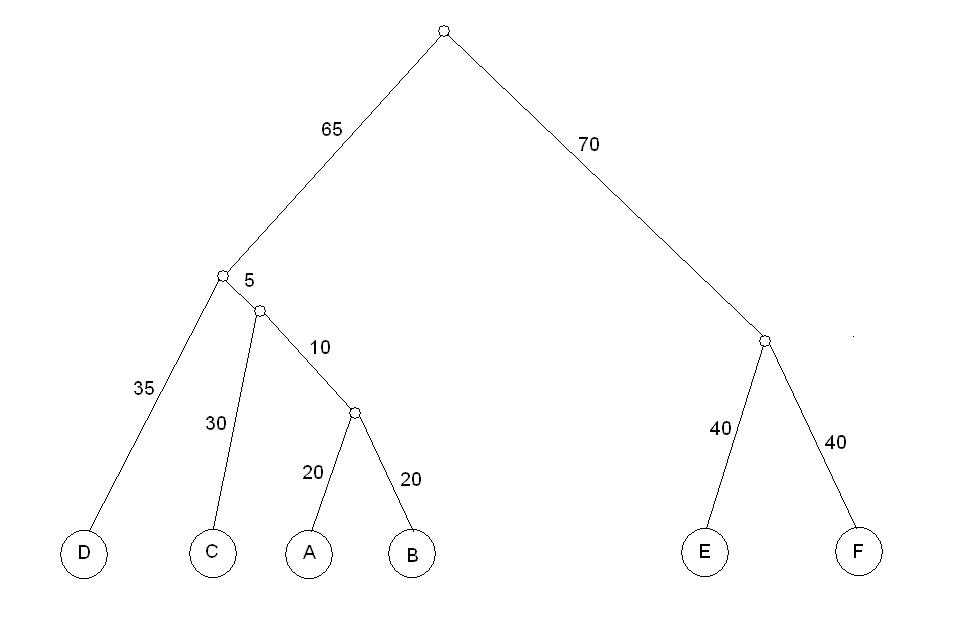
Формула, описывающая судьбу моего гена- ((((А:20,B:20):10,C:30):5,D:35):65,(E:40,F:40):70);
Изображение дерева
Так выглядит дерево, описываемое приведенной формулой.
Описание разбиения листьев дерева(если считать дерево бескорневым)
A B C D E F
. . * * * *
. . . * * *
. . . . * *
|
Получение искусственных мутантных последовательностей, соответствующим листьям и узлам дерева
Для пересчета длин ветвей была использована формула "число мутаций=длина последовательности*длина ветви\100",
а потом был составлен скрипт :
msbar ABCDEF.fasta ABCD.fasta -point 4 -count 1571 -auto
msbar ABCD.fasta D.fasta -point 4 -count 845 -auto
msbar ABCD.fasta ABC.fasta -point 4 -count 121 -auto
msbar ABC.fasta AB.fasta -point 4 -count 242 -auto
msbar AB.fasta A.fasta -point 4 -count 483 -auto
msbar AB.fasta B.fasta -point 4 -count 483 -auto
msbar ABC.fasta C.fasta -point 4 -count 725 -auto
msbar ABCDEF.fasta EF.fasta -point 4 -count 1691 -auto
msbar EF.fasta E.fasta -point 4 -count 966 -auto
msbar EF.fasta F.fasta -point 4 -count 966 -auto
|
Где название файлов соответствует содержанию (ABCDEF для общего предка, ABCD дя общего предка листьев A, B, C и D и т.д.)
Реконструкция дерева алгоритмами UPGMA, Neighbor-joining и максимального правдоподобия.
На основе предыдущего задания был создан fasta-файл, содержащий последовательности всех листьев дерева.
После этого была запущена программа fdnaml(чтобы реконструировать дерево алгоритмом максимального правдоподобия)
командой fdnaml list.fasta -ttratio 1 -auto .
Дерево, построенное данным алгоритмом выглядит так:
+----b
|
| +---------f
| +--------------------------------4
| +--3 +--------e
| | |
1--2 +------d
| |
| +------c
|
+----A
Для построения дерева алгоритмами UPGMA или Neighbor-joining были пересчитаны попарные растояния между последовательностями
программой fdnadist.
Команда fneighbor list.fdnadist -auto
Полученный файл list.fdnadist был дан на вход программе fneighbor.
Команда fneighbor list.fdnadist -auto (для реконструкции алгоритмом Neighbor-joining)
Команда fneighbor list.fdnadist -treetype u -auto -outfile list2.fneighbor (для реконструкции алгоритмом UPGMA)
В результате были получены деревья:
алгоритм Neighbor-joining
+----b
!
! +------c
3--4
! ! +------d
! +--2
! ! +-------e
! +----------------------------------1
! +---------f
!
+----A
алгоритм UPGMA
+--------A
+----1
+-2 +--------b
! !
+------------------------------------3 +-------------c
! !
--5 +---------------d
!
! +-----------------e
+----------------------------------4
+-----------------f
Таким образом видно, что алгоритм максимального правдоподобия и Neighbor-joining строят бескорневые деревья, тогда как алгоритм
UPGMA строит укоренные деревья.
Сравнение полученных деревьев
A B C D E F real Max UPGMA N-J
* * - - - - + + + +
* * * - - - + + + +
- - - - * * + + + +
|
Как видно из данной таблицы все алгоритмы выдали одинаковые деревья.
вернуться на страницу четвертого семестра
главная страница
©Подковкина Анна